From PGI
서열 데이터에서 bias를 줄이기 위한 방법
현재의 DNA sequencing 기술로 얻어낸 데이터들은 수 억 개의 염기서열을 분석하다 보니 그 과정에서 어쩔 수 없이 오차가 생기고 이상적인 데이터에 비해 약간의 차이를 가지고 있다. 만약 DNA 해독 bias로 인해 잘못된 결과가 나타난 부분인 사람의 신경물질전달자 조절자인 NCS1에서 나타났다고 생각을 해보자. 그렇다면 연구자들은 bias로 인해 실험결과는 사실과는 다르게 될 것이며, 연구자가 내린 결론 또한 잘못된 것이다. 이러한 것이 반복된다면 DNA 해독뿐만 아니라 관련된 실험 모두 신뢰를 얻지 못할 것이다.
MIT와 Broad 연구소 연구진들은 위 의 예시처럼 서열 bias로 인해 과학적, 의학적 결과물들이 신뢰를 받지 못할 수도 있기 때문에, 이러한 차이를 찾아내고 줄이기 위한 자동화 방법을 발전해 나가는 것이 중요하다고 하였다.
Relative coverage란 게놈에서 주어진 reference 염기의 coverage와 모든 reference 염기의 coverage의 비율을 의미 하는 수치 이며 이것이 1보다 높을 경우 실험자가 예상하였던 coverage 보다 더 높은 결과값이 나왔다는 것을 의미하며 1보다 낮다는 것은 예상하였던 coverage보다 낮은 결과값이 나왔다는 것을 의미한다. Relative coverage가 1에 가까울수록 이상적인 결과값이 나왔다고 할 수 있겠다. Motif란 bias와 연관이 있는 특이한 염기구성내역을 말한다.
아래 5개의 motif는 가장 흔하게 볼 수 있는 5개의 motif들이다.
• GC ≤ 10%: 중간에 GC의 비율이 10% 미만인 100염기들을 가지고 있는 200염기 구역
• GC ≥ 75%: 중간에 GC의 비율이 75% 이상인 100염기들을 가지고 있는 200염기 구역
• GC ≥ 85%: 중간에 GC의 비율이 85% 미만인 100염기들을 가지고 있는 200염기 구역
• (AT)15: 중간에 30개의 AT di-nucleotide 염기서열을 가진 130염기 구역
• G|C ≥ 80%: 중간에 G나 C의 비율이 80%이상인 30 염기서열을 가진 130염기 구역
사람은 bad promoter 라 하는 특이한 motif를 하나 더 가지고 있다.
• Bad promoters: 인간의 게놈에서 경험적으로 정의한 1000개의 200염기 간격
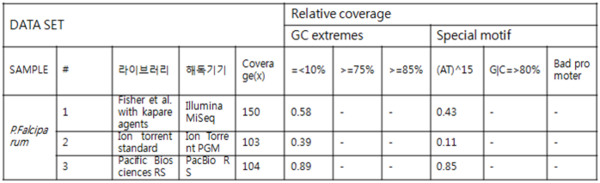
연구진들은 여러 가지 조건들을 바꾸어가며 실험을 진행하였는데, 먼저 같은 샘플에 대하여 3가지 플랫폼에서 나온 결과의 편차를 비교하였다. 결과적으로는, 대상이 된 3가지 해독 플랫폼 중 가장 오차가 적은 것은 Pacific Biosciences RS였고, 같은 샘플에 같은 플랫폼을 사용하여도 라이브러리가 다르면 bias가 달라진다는 것을 확인할 수 있었다. Pac Bio의 경우 motif 영역에서 relative coverage가 가장 높으므로 가장 편차가 적을 것이다(표1).
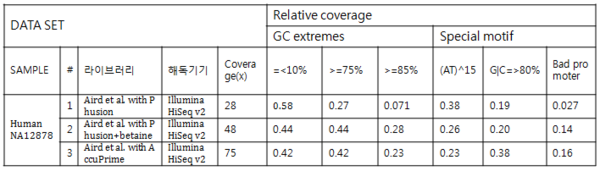
공통된 인간의 sample과 같은 해독 플랫폼 상태에서 라이브러리를 바꾸어주면 GC>=85%범위에서 relative coverage가 큰 차이를 보이는 것을 알 수 있다 이것은 라이브러리에 따라 편차가 심하게 변하는 것을 뜻하며 우리는 샘플에 맞는 적절한 라이브러리를 선택 해야 할 필요가 있다(표2).
만약 DNA를 해독 하는데 만약 편차가 무시 못 할 수준이라면 아마도 사람들은 분석결과를 믿지 못하고 그 결과를 바탕으로 만들어진 연구결과나 상용화된 제품들을 믿지 못 할 것이다. 그렇기 때문에 우리는 올바른 연구 결과와 그를 이용한 인간 생활을 발전을 위해서 bias를 가장 줄일 수 있는 실험 기기 및 실험 방법을 택하여 실험을 하며 앞으로도 bias 를 줄일 수 있는 방법을 개발 하도록 노력을 해야 할 것이다.
참고문헌
Characterizing and measuring bias in sequence data
역저자
글 : Bae.GiDong
편집 : Park.HyeonJi, Jeon.EunSook