From PGI
(Created page with "= '''침팬지 게놈에 있는 Alu 재조합에 의한 구조적 결실''' = '''''Alu ''element'''는 '''S'''hort'''IN'''terspersed '''E'''lement(SINE)의 그룹...") |
|||
| Line 10: | Line 10: | ||
| + | |||
=== 침팬지 특이적인 ARMD 현상 === | === 침팬지 특이적인 ARMD 현상 === | ||
| − | |||
| − | |||
| − | + | panTro1에서 총 1,538개의 ARMD loci들이 검색되었으나, 연구도중 어셈블리가 향상된 panTro2가 확인되어 panTro1과 panTro2와의 비교를 통해, 258개가 panTro1에서 잘못된 것임을 밝혔다. 최종적으로 RepeatMasker를 사용하여 513개를 제외시켰다. 남은 1,025개의 loci들은 PCR 분석, ''Alu ''element의 TSD분석, 그리고 인간, 침팬지, 레서스 원숭이(''rhesus macaque'')의 유사 염기서열들을 정렬하여, 663개의 침팬지 특이적인 ARMD loci를 확인하였다. | |
| − | |||
| − | |||
| − | + | [[File:Alu_element_01.png|center|500px|Alu element 01.png]] | |
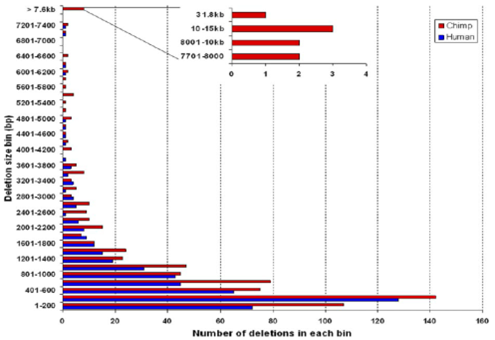
| − | [[ | + | 침팬지 특이적인 ARMD에서 결실된 DNA 서열은 평균 1,164bp의 길이로, 이전에 연구된 인간 특이적인 ARMD와 유사한 경향을 보이며, 약 68%가 1kb 미만이다. 하지만, 인간 특이적인 ARMD에서는 관찰되지 않았던 7.3kb이상의 결실이 침팬지에서는 10개나 발견되었다. |
| + | |||
| + | |||
| + | |||
| + | [[File:Alu element 02.png|center|500px|Alu element 02.png]] | ||
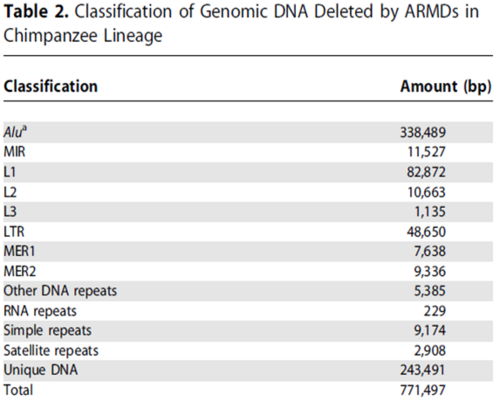
결실된 gDNA 서열의 약 70%는 중복되는 요소들을 포함하고 있었고, 이 중 ''Alu ''element가 약 64%를 차지하였다. 침팬지 ARMD에 의해 결실된 DNA 서열의 길이는 같은 기간 일어난 인간 ARMD보다 2배로 나타났고, 유사 loci를 비교했을 때, 약 46%의 ARMD가 알려져 있거나 참조서열의 유전자에 위치해 있었다. 그 중 5개 ARMD에 의해 6개 유전자 내에서 13개의 엑손 영역의 결실을 야기했다. 또한 2개의 ARMD는 ''NBR2''와 ''HTR3D''라는 기능 유전자의 엑손에서 결실을 일으켰으며, 나머지 3개는 기능이 밝혀지지 않은 4개의 유전자들에서 엑손 결실을 야기하였다. | 결실된 gDNA 서열의 약 70%는 중복되는 요소들을 포함하고 있었고, 이 중 ''Alu ''element가 약 64%를 차지하였다. 침팬지 ARMD에 의해 결실된 DNA 서열의 길이는 같은 기간 일어난 인간 ARMD보다 2배로 나타났고, 유사 loci를 비교했을 때, 약 46%의 ARMD가 알려져 있거나 참조서열의 유전자에 위치해 있었다. 그 중 5개 ARMD에 의해 6개 유전자 내에서 13개의 엑손 영역의 결실을 야기했다. 또한 2개의 ARMD는 ''NBR2''와 ''HTR3D''라는 기능 유전자의 엑손에서 결실을 일으켰으며, 나머지 3개는 기능이 밝혀지지 않은 4개의 유전자들에서 엑손 결실을 야기하였다. | ||
| − | |||
| − | |||
=== ARMD “핫스팟” === | === ARMD “핫스팟” === | ||
Latest revision as of 10:45, 11 October 2022
Contents
침팬지 게놈에 있는 Alu 재조합에 의한 구조적 결실
Alu element는 ShortINterspersed Element(SINE)의 그룹 중 하나이며, 약 6,500만년 전에 나타나 현재까지 영장류 게놈에 약 120만 이상의 copy를 차지하고 있다. Alu는 left와 right 단위체로 구성되어 있다. 각각의 단위체들은 7SL-RNA로부터 독립적으로 유래되었으며, 두 단위체가 융합되어 dimeric Alu로 나타났다. Alu의 subfamily들 간의 염기서열 유사도는 70-99.7%로 상당히 높다. Alu 재조합에 의해 일어나는 구조적 결실(ARMD; Alu Recombination-Mediated Deletion)은 같은 염색체상의 두 Alu 사이에 일어나는 염색체 내 재조합 또는 감수 분열 동안 염색 분체가 될 때 일어나는 염색체 간 재조합에 의해 발생될 수 있다.
침팬지와 사람 유전체들의 비교 분석을 통해 663개의 침팬지 특이적인 ARMD를 동정하였다. 침팬지에 특이적인 ARMD를 통해 게놈에서 총 771kb가 결실되었음을 밝혔다.
침팬지 특이적인 ARMD 현상
panTro1에서 총 1,538개의 ARMD loci들이 검색되었으나, 연구도중 어셈블리가 향상된 panTro2가 확인되어 panTro1과 panTro2와의 비교를 통해, 258개가 panTro1에서 잘못된 것임을 밝혔다. 최종적으로 RepeatMasker를 사용하여 513개를 제외시켰다. 남은 1,025개의 loci들은 PCR 분석, Alu element의 TSD분석, 그리고 인간, 침팬지, 레서스 원숭이(rhesus macaque)의 유사 염기서열들을 정렬하여, 663개의 침팬지 특이적인 ARMD loci를 확인하였다.
침팬지 특이적인 ARMD에서 결실된 DNA 서열은 평균 1,164bp의 길이로, 이전에 연구된 인간 특이적인 ARMD와 유사한 경향을 보이며, 약 68%가 1kb 미만이다. 하지만, 인간 특이적인 ARMD에서는 관찰되지 않았던 7.3kb이상의 결실이 침팬지에서는 10개나 발견되었다.
결실된 gDNA 서열의 약 70%는 중복되는 요소들을 포함하고 있었고, 이 중 Alu element가 약 64%를 차지하였다. 침팬지 ARMD에 의해 결실된 DNA 서열의 길이는 같은 기간 일어난 인간 ARMD보다 2배로 나타났고, 유사 loci를 비교했을 때, 약 46%의 ARMD가 알려져 있거나 참조서열의 유전자에 위치해 있었다. 그 중 5개 ARMD에 의해 6개 유전자 내에서 13개의 엑손 영역의 결실을 야기했다. 또한 2개의 ARMD는 NBR2와 HTR3D라는 기능 유전자의 엑손에서 결실을 일으켰으며, 나머지 3개는 기능이 밝혀지지 않은 4개의 유전자들에서 엑손 결실을 야기하였다.
ARMD “핫스팟”
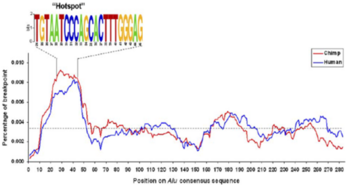
재조합 지점을 확인하기 위해, 침팬지 게놈에서 ARMD가 일어난 Alu와 인간에서 변하지 않은 2개의 Alu들을 정렬하였다. 두 개의 Alu 사이에 재조합이 일어나는 범위는 평균 20bp였다. Alu 들의 보존된 서열들을 정렬하였을 때, 24~45bp 사이에 위치한 재조합 “핫 스팟”을 확인하였으며, 이 염기서열은 Alu element들간에 가장 보존된 지역에 위치한다는 것을 알아냈다. 또한 핫스팟 서열은 recBC에 의존된 재조합을 활성시키는 것으로 알려진 원핵 chi 서열을 포함하고 있었다.
대부분의 Alu는 영장류 게놈 상의 GC 함량이 높은 지역에 위치한다. 그리고 Alu 역시 높은 GC 비율을 지니고 있다. 침팬지 특이적인 ARMD loci의 주변을 확인하기 위해, 양쪽 10kb씩 분석한 결과, 인간 특이적인 ARMD와 유사하게 GC가 많은 지역에 발생하는 경향을 보였다. 또한 각각의 chimeric Alu 들의 양쪽 지역 2Mb씩 총 4Mb를 확인하여 유전 밀도를 추산하여, 전반적인 분포는 유전자가 많은 지역에 편향되어 있다고 볼 수 있다.
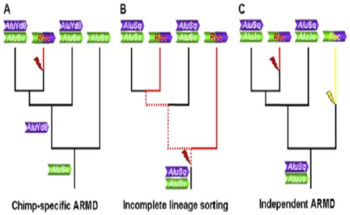
Computational 분석과 wet-bench 분석으로 663개의 침팬지 특이적인 ARMD loci를 확인했을 때, 정확하게 분석되지 않는 16개의 loci를 발견했다. 16개 중 11개는 인간과 고릴라 게놈에 두 개의 Alu가 존재하지만, 침팬지와 오랑우탄 게놈의 유사 지역에는 하나의 Alu 가 존재하고 있었다. 분석 결과 11개 중에 5개는 침팬지에 특이적인 ARMD로써 인간, 침팬지, 고릴라의 공통조상과 오랑우탄과의 분기 이후에 host 게놈으로 침팬지의 ARMD를 일으킨 두 Alu element 중 하나가 삽입되어 있었다. 침팬지와 오랑우탄의 게놈에서 유사 loci는 다른 시기에 두 가지 ARMD가 발생했음을 보여주는 근거가 된다.
특이적인 ARMD의 연관성 수준
인간과 침팬지의 게놈은 전반적으로 유사성이 높지만, 초기 분화 이후에 삽입과 결실, 반전(inversion), 중복(duplication)과 같은 유전적 변화에 때문에, 미묘한 차이를 보이게 되었다. 기전은 유사하지만, 일어난 횟수와 결실 부위의 크기는 상당히 다르다. 이 두 종의 ARMD 사이에 차이점은 분기 이후에 인간의 인구가 감소되고, 유전적 다양성이 감소되었다는 것이다. 이는 인간보다 높은 침팬지 특이적인 ARMD의 다형성 수준에 의해 뒷받침 된다.
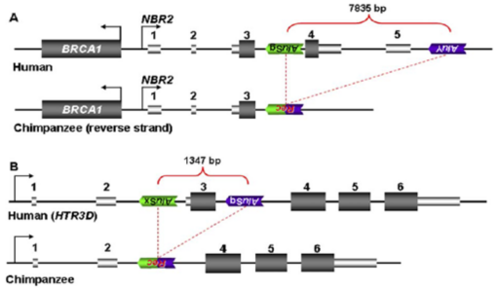
앞서 언급한 인간 게놈에서 기능적인 유전자(NBR2와 HTR3D)안에 엑손을 deletion 시킨 두 개의 침팬지 특이적인 ARMD는 인간과 침팬지 사이에 표현형 적 차이를 만드는 데 기여했다는 증거가 된다.
NBR2 유전자는 인간 게놈에서 염색체 17번 BRCA1 유전자 바로 옆에 위치하며, BRACA1과 프로모터(promoter)를 공유한다. 인간의 경우, 인트론에 결합하는 비 조직의 특이적인 단백질 복합체에 의해 억제된다. 하지만 침팬지에서는 인트론과 3’ 지역 사이에 엑손 결실을 일으키는 ARMD가 발생하였다. 이러한 ARMD는 침팬지 게놈에서 억제제의 존재 유무에 영향을 받지 않고, 잠재적으로 발현이 저해되는 결과를 야기했다.
HTR3D는 5-HT3 세로토닌 수용체 유전자 그룹에 속해 있으며, 위장 기관 내 세로토닌에 관여를 한다. 침팬지 게놈에서 HTR3D 유전자의 첫 번째 코딩 엑손이 ARMD에 의해 결실되었다. 이는 인간과 침팬지 사이에 생리학적 변화에 영향을 준다고 생각된다.
맺음말
Retrotransposon에 매개되는 유전적 재정렬은 유전체 내의 종 특이적 변화의 중요한 요소 중 하나로 작용한다. 인간과 침팬지 사이의 ARMD 비교 연구는 생물학적 차이에 매우 중요한 역할을 하는 것을 알 수 있다. 또한, 침팬지 특이적인 AMRD 과정은 인간과의 분기 이후에 새로운 Alu 삽입에 의해 일어나는 유전적 확장에서 균형을 유지하게 한다. 침팬지의 663개 ARMD와 유사한 loci는 인간에서도 불안정한 구조에 해당되는데, 침팬지에서 일어난 ARMD가 인간과 다른 유인원에게서도 일어날 가능성이 있음을 보여주기 때문이다.
참고문헌
Alu Recombination-Mediated Structural Deletions in the Chimpanzee Genome.
http://www.plosgenetics.org/article/info%3Adoi%2F10.1371%2Fjournal.pgen.0030184
역저자
글 : Han. KyuDong(kyudong.han@gmail.com)
편집 : Park. HyeonJi(hyeonjipark09@gmail.com)
키워드 : Retrotransposon, HTR3D, NBR2, Alu element, ARMD(Alu Recombination-Mediated Deletion) 등