From PGI
(Created page with "= '''조직 재생에 관련된 붉은점영원(''Natophthalmus viridescens'')의 전사체 특징''' = center|250px ...") |
|||
| Line 1: | Line 1: | ||
| + | |||
= '''조직 재생에 관련된 붉은점영원(''Natophthalmus viridescens'')의 전사체 특징''' = | = '''조직 재생에 관련된 붉은점영원(''Natophthalmus viridescens'')의 전사체 특징''' = | ||
| − | |||
| − | |||
| − | + | [[File:GT_REVIEW_201305_Newt_01.png|center|250px|GT REVIEW 201305 Newt 01.png]] 그림 1 ''Notophthalmus viridescens'' | |
| + | |||
| + | |||
| + | |||
| + | '''''Natophthalmus viridescens'''''는 양서류이자 유미류로서 재생의학 연구에 탁월한 모델로 알려져 있다. 그러나 재생 과정에 대한 메커니즘은 부정확한 annotation과 발현되는 염기 서열의 정보 부족으로 인해 밝혀지지 않았다. 방대한 크기의 게놈 사이즈와 유사 종의 참조 서열의 부재는 유미류 게놈의 조립(assembly)에 큰 장애물이다. | ||
| − | |||
| − | |||
=== 붉은점영원의 전사체 해독 === | === 붉은점영원의 전사체 해독 === | ||
| − | |||
| − | |||
| − | + | 붉은점영원의 전사체를 해독하기 위해서, 심장에서 추출한 cDNA를 높은 배수로 라이브러리를 제작하였다. 또한 454 플랫폼을 이용하여 310bp의 read를 해독(pyrosequencing)하여 80,7466개의 전사체를 확인하였다. 또 수정체 적출술을 통해 추출한지 4~8일 지난 샘플의 홍체에서 추출한 cDNA를 일루미나 read(paired end)로 679,816,626개의 전사체를 확인하였다. | |
| + | |||
| + | |||
| + | |||
| + | 해독된 read의 효과적인 조립을 위해 4가지 다른 방법을 시도하였다. | ||
| + | |||
| + | |||
| + | |||
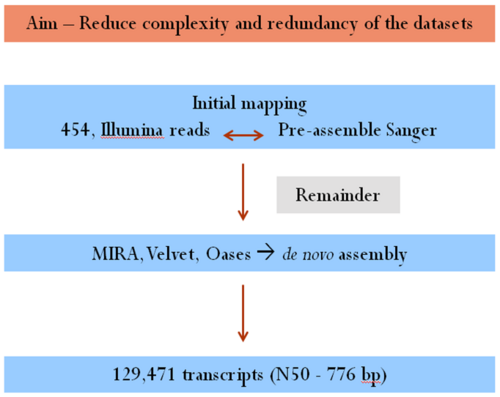
| + | [[File:GT REVIEW 201305 Newt 02.png|center|500px|GT REVIEW 201305 Newt 02.png]] 그림 2 첫 번째 방법, 데이터의 복잡성과 중복성을 줄이기 위함 | ||
| − | |||
| − | |||
| − | [ | + | [그림 2]는 첫 번째 방법으로, 데이터의 복잡성과 중복성을 줄이기 위해, 454와 일루미나로부터 나온 초기 데이터를 미리 제작된 생어 read와 비교한 것이다. |
| − | |||
| − | |||
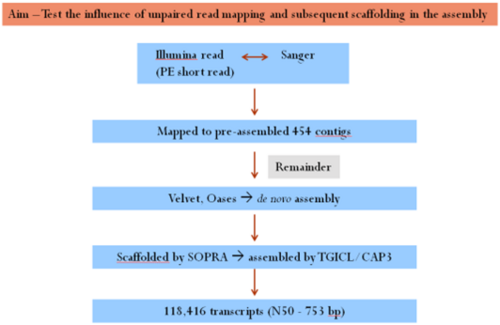
| − | + | [[File:GT REVIEW 201305 Newt 03.png|center|500px|GT REVIEW 201305 Newt 03.png]] 그림 3 쌍을 이루지 않는 read의 매핑이 이후 조립에 영향을 끼치는 지 확인 | |
| − | |||
| − | |||
| − | [그림 3]은 두 번째 방법으로, 첫 번째와 같이 모든 일루미나 read를 미리 조립한 생어 read에 매핑하고, 미리 조립된 454 contig를 매핑하는 방법이다. 이는 쌍을 이루지 않는 read의 매핑이 이후 조립에 영향을 끼치는 지 확인한 것이다. | + | [그림 3]은 두 번째 방법으로, 첫 번째와 같이 모든 일루미나 read를 미리 조립한 생어 read에 매핑하고, 미리 조립된 454 contig를 매핑하는 방법이다. 이는 쌍을 이루지 않는 read의 매핑이 이후 조립에 영향을 끼치는 지 확인한 것이다. |
| − | |||
| − | |||
| − | + | [[File:GT REVIEW 201305 Newt 04.png|center|500px|GT REVIEW 201305 Newt 04.png]] 그림 4 참조 데이터가 조립에 미치는 영향을 확인 | |
| − | |||
| − | |||
| − | [ | + | [그림 4]는 세 번째 방법으로, 참조 데이터가 조립에 미치는 영향을 확인한 것이다. 유사 종의 결과 데이터를 이용하여 매핑을 하고, 새로 조립을 하였다. 이후 이전 데이터인 생어 데이터를 추가로 삽입하여, 조립에 미치는 영향을 확인하는 방법이다. |
| − | |||
| − | |||
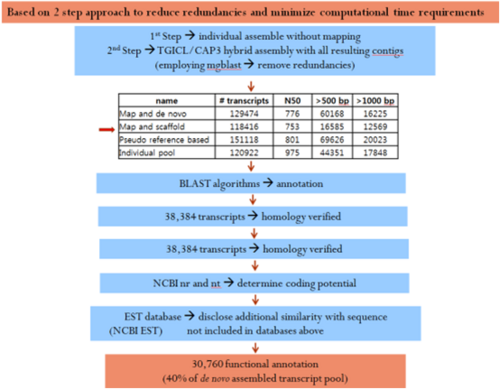
| − | + | [[File:GT REVIEW 201305 Newt 05.png|center|500px|GT REVIEW 201305 Newt 05.png]] 그림 5 중복성을 줄이고, 분석 시간을 줄이기 위한 방법 | |
| + | |||
| + | |||
| + | |||
| + | [그림 5]는 중복성을 줄이고, 분석시간을 줄이기 위한 네 번째 방법으로, 최종 참조 전사체가 두가지 단계의 전략을 이용하여 제작된 것이다. 첫 번째 단계는 각각의 서열이 독립적으로 매핑되지 않고 조립되었고, 두 번째 단계에서는 중복성을 줄이기 위해 모든 contig 결과를 바탕으로 TGICL과 CAP3을 포함한 BLAST를 이용하여 hybrid 조립이 진행되었다. | ||
| + | |||
| + | |||
| + | |||
| + | === 높은 품질의 전사체 annotation 및 생정보 분석 === | ||
| − | |||
| − | |||
전사체 annotation은 BLAST 알고리즘의 상동성 조사를 통해 이루어졌다. 총 38,384개 전사체가 하나 이상의 상동성을 가지고 있었는데, 이미 알려진 단백질 발현 서열과의 유사성 비교를 위해 NCBI 데이터를 검색해 보았다. | 전사체 annotation은 BLAST 알고리즘의 상동성 조사를 통해 이루어졌다. 총 38,384개 전사체가 하나 이상의 상동성을 가지고 있었는데, 이미 알려진 단백질 발현 서열과의 유사성 비교를 위해 NCBI 데이터를 검색해 보았다. | ||
| − | |||
| − | |||
| − | + | 총 30,760개의 기능을 annotation한 것을 확인하였는데, 새로이 조립된 전사체의 40%에 해당하는 양이다. 여기에 전사체 길이와 진핵생물의 체세포의 핵의 반수체의 DNA 양을 뜻하는 c-value에 따른 염기서열의 annotation 비율을 비교하는 분석도 추가로 이루어졌다. 이러한 분석 결과, 발현이 안 되는 짧은 서열과 잠재적 발현이 있을 서열에 대한 구별이 가능한 것으로 드러났다. | |
| + | |||
| + | |||
| + | |||
| + | 붉은점영원의 참조 전사체 제작의 가장 큰 목적은 앞으로의 NGS와 대량의 단백질학(proteomics) 방법을 위한 통합적인 데이터를 확립하는 데 있다. 그러므로 이번 annotation의 목적은 많은 양 보다는 높은 질을 우선으로 하였으며, 참조 전사체의 해독 배수는 일반적인 전사 신호 경로와 알려진 유전자 그룹들로 추산되었는데, 특이한 점은 80% 이상에서 인간의 p53과 TGF beta 경로와 유사한 그룹이 발견된 것이다. | ||
| − | |||
| − | |||
이번 연구로 알게 된 붉은점영원의 전사체에서 발현 잠재성을 확인하기 위해, reverse-phase nano-LC-MS/MS와 같은 다양한 질량분석법이 진행되었다. 심장, 수정체, 꼬리, 간, 비장 등에서 재생 시간 별 단백질들을 추출하였으며, SILAC(Stable isotope labeling by amino acids in cell culture; 세포 배양 시 서로 다른 조건의 시료 중 단백질을 정량 분석하는 데 사용하는 방법) 기반의 조직에서는 데이터의 품질을 크게 증가시켰다. 또한 LC-MS/MS를 통해 발견된 단백질들은 붉은점영원의 전사체 내에 모든 발현 가능한 염기서열을 이용하여 비교 분석하였다. 그 결과, 14,471개의 전사체에 상응하는 55,605개 단백질이 확인되었으며, 이는 120,922개의 중복되지 않은 전사체의 약 12%에 해당하는 양이다. | 이번 연구로 알게 된 붉은점영원의 전사체에서 발현 잠재성을 확인하기 위해, reverse-phase nano-LC-MS/MS와 같은 다양한 질량분석법이 진행되었다. 심장, 수정체, 꼬리, 간, 비장 등에서 재생 시간 별 단백질들을 추출하였으며, SILAC(Stable isotope labeling by amino acids in cell culture; 세포 배양 시 서로 다른 조건의 시료 중 단백질을 정량 분석하는 데 사용하는 방법) 기반의 조직에서는 데이터의 품질을 크게 증가시켰다. 또한 LC-MS/MS를 통해 발견된 단백질들은 붉은점영원의 전사체 내에 모든 발현 가능한 염기서열을 이용하여 비교 분석하였다. 그 결과, 14,471개의 전사체에 상응하는 55,605개 단백질이 확인되었으며, 이는 120,922개의 중복되지 않은 전사체의 약 12%에 해당하는 양이다. | ||
| − | + | ||
=== 유미류 특이적인 단백질의 동정 === | === 유미류 특이적인 단백질의 동정 === | ||
| − | |||
| − | |||
| − | + | 조직 재생과 같은 종 특이적인 생물학적 현상에 연관된 유전자를 규명하기 위해, 유미류가 아닌 종에서는 나타나지 않는 새로운 단백질을 발현하는 전사체들을 분석하였다. 잘못된 서열 구조를 피하기 위해, 질량분석법으로 확인된 단백질 발현 유전자들만 그룹화하였고, 826개의 단백질 발현 전사체가 공개된 데이터에서 하나도 나타나지 않음을 확인하였다. | |
| + | |||
| + | |||
| + | |||
| + | 유미류 특이적인 전사체에 위치한 도메인은 액티빈(activin) 수용체를 포함하고 있는데, 이는 사람의 성장인자 변형 수용체(A6MIV6_HUMAN)에서도 발견되는 도메인이다. 다른 예로는, 파신(fascin) 수용체를 들 수 있는데, 구조 특이적으로 액틴 단백질을 교차 결합시키는 번들링(bundling)을 유도하는 도메인이다. | ||
| − | |||
| − | |||
=== 맺음말 === | === 맺음말 === | ||
| − | |||
| − | |||
| − | + | 대용량 해독은 복잡한 생물체의 전체 게놈을 해독하는 데 있어서, 필수적인 도구가 되었다. 해독 가격의 하락으로 인해, 더욱더 가속화되고 있으나, De novo 해독은 아직까지 가격이 높다. 게놈 사이즈가 크거나 복잡한 생물체의 경우, 포괄적인 유전적 분석에 이용 가능성이 높지만, 이루어 지지 않는 경우가 많다. 그런 점에서, 이번 연구는 재생 능력이 뛰어난 붉은점영원의 전사체를 연구하여 앞으로 파생될 연구 및 게놈 프로젝트의 발판을 마련한 것이다. | |
| + | |||
| + | |||
=== 참고문헌 === | === 참고문헌 === | ||
| − | + | ||
A de novo assembly of the newt transcriptome combined with proteomic validation identifies new protein families expressed during tissue regeneration | A de novo assembly of the newt transcriptome combined with proteomic validation identifies new protein families expressed during tissue regeneration | ||
| − | http://genomebiology.com/2013/14/2/R16/abstract | + | [http://genomebiology.com/2013/14/2/R16/abstract http://genomebiology.com/2013/14/2/R16/abstract] |
KEGG for integration and interpretation of large-scale molecular data sets. | KEGG for integration and interpretation of large-scale molecular data sets. | ||
| − | http://nar.oxfordjournals.org/content/early/2011/11/10/nar.gkr988.full | + | [http://nar.oxfordjournals.org/content/early/2011/11/10/nar.gkr988.full http://nar.oxfordjournals.org/content/early/2011/11/10/nar.gkr988.full] |
Spiked-in Pulsed in Vivo Labeling Identifies a New Member of the CCN Family in Regenerating Newt Hearts. | Spiked-in Pulsed in Vivo Labeling Identifies a New Member of the CCN Family in Regenerating Newt Hearts. | ||
| − | http://pubs.acs.org/doi/abs/10.1021/pr300521p?mi=z48nb4& | + | [http://pubs.acs.org/doi/abs/10.1021/pr300521p?mi=z48nb4&af=R&pageSize=20&searchText=ageing http://pubs.acs.org/doi/abs/10.1021/pr300521p?mi=z48nb4&af=R&pageSize=20&searchText=ageing] |
| + | |||
| − | |||
=== 역저자 === | === 역저자 === | ||
| − | |||
| − | |||
| − | + | 글 : Son.BongJun | |
| − | + | 편집 : Park.HyeonJi, Ahn.Kung | |
| − | + | 키워드 : 붉은점영원(''Natophthalmus viridescens''), 유미류(Urodela), annotation, assembly, activin, fascin 등 | |
Latest revision as of 10:40, 11 October 2022
Contents
조직 재생에 관련된 붉은점영원(Natophthalmus viridescens)의 전사체 특징
그림 1 Notophthalmus viridescens
Natophthalmus viridescens는 양서류이자 유미류로서 재생의학 연구에 탁월한 모델로 알려져 있다. 그러나 재생 과정에 대한 메커니즘은 부정확한 annotation과 발현되는 염기 서열의 정보 부족으로 인해 밝혀지지 않았다. 방대한 크기의 게놈 사이즈와 유사 종의 참조 서열의 부재는 유미류 게놈의 조립(assembly)에 큰 장애물이다.
붉은점영원의 전사체 해독
붉은점영원의 전사체를 해독하기 위해서, 심장에서 추출한 cDNA를 높은 배수로 라이브러리를 제작하였다. 또한 454 플랫폼을 이용하여 310bp의 read를 해독(pyrosequencing)하여 80,7466개의 전사체를 확인하였다. 또 수정체 적출술을 통해 추출한지 4~8일 지난 샘플의 홍체에서 추출한 cDNA를 일루미나 read(paired end)로 679,816,626개의 전사체를 확인하였다.
해독된 read의 효과적인 조립을 위해 4가지 다른 방법을 시도하였다.
그림 2 첫 번째 방법, 데이터의 복잡성과 중복성을 줄이기 위함
[그림 2]는 첫 번째 방법으로, 데이터의 복잡성과 중복성을 줄이기 위해, 454와 일루미나로부터 나온 초기 데이터를 미리 제작된 생어 read와 비교한 것이다.
그림 3 쌍을 이루지 않는 read의 매핑이 이후 조립에 영향을 끼치는 지 확인
[그림 3]은 두 번째 방법으로, 첫 번째와 같이 모든 일루미나 read를 미리 조립한 생어 read에 매핑하고, 미리 조립된 454 contig를 매핑하는 방법이다. 이는 쌍을 이루지 않는 read의 매핑이 이후 조립에 영향을 끼치는 지 확인한 것이다.
그림 4 참조 데이터가 조립에 미치는 영향을 확인
[그림 4]는 세 번째 방법으로, 참조 데이터가 조립에 미치는 영향을 확인한 것이다. 유사 종의 결과 데이터를 이용하여 매핑을 하고, 새로 조립을 하였다. 이후 이전 데이터인 생어 데이터를 추가로 삽입하여, 조립에 미치는 영향을 확인하는 방법이다.
그림 5 중복성을 줄이고, 분석 시간을 줄이기 위한 방법
[그림 5]는 중복성을 줄이고, 분석시간을 줄이기 위한 네 번째 방법으로, 최종 참조 전사체가 두가지 단계의 전략을 이용하여 제작된 것이다. 첫 번째 단계는 각각의 서열이 독립적으로 매핑되지 않고 조립되었고, 두 번째 단계에서는 중복성을 줄이기 위해 모든 contig 결과를 바탕으로 TGICL과 CAP3을 포함한 BLAST를 이용하여 hybrid 조립이 진행되었다.
높은 품질의 전사체 annotation 및 생정보 분석
전사체 annotation은 BLAST 알고리즘의 상동성 조사를 통해 이루어졌다. 총 38,384개 전사체가 하나 이상의 상동성을 가지고 있었는데, 이미 알려진 단백질 발현 서열과의 유사성 비교를 위해 NCBI 데이터를 검색해 보았다.
총 30,760개의 기능을 annotation한 것을 확인하였는데, 새로이 조립된 전사체의 40%에 해당하는 양이다. 여기에 전사체 길이와 진핵생물의 체세포의 핵의 반수체의 DNA 양을 뜻하는 c-value에 따른 염기서열의 annotation 비율을 비교하는 분석도 추가로 이루어졌다. 이러한 분석 결과, 발현이 안 되는 짧은 서열과 잠재적 발현이 있을 서열에 대한 구별이 가능한 것으로 드러났다.
붉은점영원의 참조 전사체 제작의 가장 큰 목적은 앞으로의 NGS와 대량의 단백질학(proteomics) 방법을 위한 통합적인 데이터를 확립하는 데 있다. 그러므로 이번 annotation의 목적은 많은 양 보다는 높은 질을 우선으로 하였으며, 참조 전사체의 해독 배수는 일반적인 전사 신호 경로와 알려진 유전자 그룹들로 추산되었는데, 특이한 점은 80% 이상에서 인간의 p53과 TGF beta 경로와 유사한 그룹이 발견된 것이다.
이번 연구로 알게 된 붉은점영원의 전사체에서 발현 잠재성을 확인하기 위해, reverse-phase nano-LC-MS/MS와 같은 다양한 질량분석법이 진행되었다. 심장, 수정체, 꼬리, 간, 비장 등에서 재생 시간 별 단백질들을 추출하였으며, SILAC(Stable isotope labeling by amino acids in cell culture; 세포 배양 시 서로 다른 조건의 시료 중 단백질을 정량 분석하는 데 사용하는 방법) 기반의 조직에서는 데이터의 품질을 크게 증가시켰다. 또한 LC-MS/MS를 통해 발견된 단백질들은 붉은점영원의 전사체 내에 모든 발현 가능한 염기서열을 이용하여 비교 분석하였다. 그 결과, 14,471개의 전사체에 상응하는 55,605개 단백질이 확인되었으며, 이는 120,922개의 중복되지 않은 전사체의 약 12%에 해당하는 양이다.
유미류 특이적인 단백질의 동정
조직 재생과 같은 종 특이적인 생물학적 현상에 연관된 유전자를 규명하기 위해, 유미류가 아닌 종에서는 나타나지 않는 새로운 단백질을 발현하는 전사체들을 분석하였다. 잘못된 서열 구조를 피하기 위해, 질량분석법으로 확인된 단백질 발현 유전자들만 그룹화하였고, 826개의 단백질 발현 전사체가 공개된 데이터에서 하나도 나타나지 않음을 확인하였다.
유미류 특이적인 전사체에 위치한 도메인은 액티빈(activin) 수용체를 포함하고 있는데, 이는 사람의 성장인자 변형 수용체(A6MIV6_HUMAN)에서도 발견되는 도메인이다. 다른 예로는, 파신(fascin) 수용체를 들 수 있는데, 구조 특이적으로 액틴 단백질을 교차 결합시키는 번들링(bundling)을 유도하는 도메인이다.
맺음말
대용량 해독은 복잡한 생물체의 전체 게놈을 해독하는 데 있어서, 필수적인 도구가 되었다. 해독 가격의 하락으로 인해, 더욱더 가속화되고 있으나, De novo 해독은 아직까지 가격이 높다. 게놈 사이즈가 크거나 복잡한 생물체의 경우, 포괄적인 유전적 분석에 이용 가능성이 높지만, 이루어 지지 않는 경우가 많다. 그런 점에서, 이번 연구는 재생 능력이 뛰어난 붉은점영원의 전사체를 연구하여 앞으로 파생될 연구 및 게놈 프로젝트의 발판을 마련한 것이다.
참고문헌
A de novo assembly of the newt transcriptome combined with proteomic validation identifies new protein families expressed during tissue regeneration
http://genomebiology.com/2013/14/2/R16/abstract
KEGG for integration and interpretation of large-scale molecular data sets.
http://nar.oxfordjournals.org/content/early/2011/11/10/nar.gkr988.full
Spiked-in Pulsed in Vivo Labeling Identifies a New Member of the CCN Family in Regenerating Newt Hearts.
http://pubs.acs.org/doi/abs/10.1021/pr300521p?mi=z48nb4&af=R&pageSize=20&searchText=ageing
역저자
글 : Son.BongJun
편집 : Park.HyeonJi, Ahn.Kung
키워드 : 붉은점영원(Natophthalmus viridescens), 유미류(Urodela), annotation, assembly, activin, fascin 등