From PGI
(Created page with "= ''' NGS 기술의 현재: NGS 기술의 임상 적용 Yes or No?''' = 인간게놈프로젝트(human genome project, HGP)의 완료로 30억 쌍의 인간 DNA...") |
|||
| Line 1: | Line 1: | ||
| − | |||
| − | | + | = ''' NGS 기술의 현재: NGS 기술의 임상 적용 Yes or No?''' = |
| − | + | 인간게놈프로젝트(human genome project, HGP)의 완료로 30억 쌍의 인간 DNA 염기가 해독됨에 따라, 인간 게놈으로부터 얻은 정보를 진단과 치료에 활용하기 위한 노력이 계속되고 있다. 드물긴 하지만, 현재도 유전 질환의 진단에 활용하고, 유전형에 맞추어 치료방법을 선택하는 몇몇 사례가 보고되었다. 이는 post-HGP와 대용량 데이터 처리기술의 발달이 이뤄 낸 성과이다. | |
| − | + | | |
| − | + | 차세대 게놈 분석기가 최초로 도입된 2005년 이래, 끊임없는 발전을 거듭하여 2012년 현재는 데이터 생산량이 확연히 늘고, 에러율이 줄어들었을 뿐 아니라, 다양한 사이즈의 라이브러리 제작이 가능하게 되었다. 결과, reference 서열이 없이도 분석 가능한 de novo sequencing, 코딩 영역만 해독하는 exome sequencing, 유전자의 발현 변화를 볼 수 있는 transcriptome profiling, 질병의 진행여부 등 환경 변화에 따른 메틸화 정도를 알 수 있는 methylation profiling, 토양이나 물 등의 환경 시료를 이용하는 metagenome 연구까지도 NGS를 이용하여 가능하게 되었다. 게다가, 연구를 넘어서 진단에까지 이용되고 있는데, 특히, NGS를 이용한 암진단 사례가 급속히 늘고 있다. | |
| − | | + | |
| − | + | Welch 등은 전장유전체 분석을 이용하여 oncogene의 fusion이 일어났음을 알아내어 stem cell plantation 치료 대신 화학요법 치료로 방향을 전환할 수 있었음을 보고하였다. | |
| − | | + | |
| − | + | 또한 전장유전체 분석을 이용하여 환자의 정상 피부와 골수(leukemia)를 이용하여 초기 유방암과 자궁암의 진단에 활용한 예가 있다. 이 환자의 경우, 피부 세포에 TP53 유전자의 exon 7-9의 novel한 3Kb heterozygous deletion이 있으며, leukemia 세포에 같은 영역의 homozygous deletion이 있음을 시퀀싱을 통해 발견하였고, 이 변이에 의해 암에 걸렸을 확률이 높다는 것을 발견하였다. | |
| − | | + | |
| − | + | Mellman 등은 Ion Torrent PGM machine을 이용하여 전염성이 강한 Shiga toxin을 생산하는 ''E.coli O104:H4''를 10일 만에 동정하여 발표함으로써 독일에서 발생한 식중독 원인균을 찾아내는 데 크게 공헌하여 화제가 된 바 있다. | |
| − | | + | |
| − | + | 위와 같이 진단 등에 NGS를 활용할 수 있게 된 이유는 NGS 기술이 끊임없이 향상되고, 시퀀싱 단가가 줄어들었기 때문이다. 특히 MiSeq, 454 GS Junior, Ion Torrent PGM과 같은 소형 해독기(bench top sequencer)의 출현으로 인해 가능해 진 것이다. | |
| − | + | | |
| − | + | | |
| − | <br> | + | <br/> 다음 표는 '''bench top sequencer들의 장점과 단점'''을 요약한 것이다. |
| − | + | | |
| − | + | [[File:NGS 임상적용.PNG|center|600px|NGS 임상적용.PNG]] | |
| − | + | 고전적으로 Sanger sequencing과 PCR 방법은 암 및 감염성 질환의 유전 변이를 진단하는 도구로 이용되어 왔으며, 그 예로 Myraid Genetic Laboratory사의 BRACAnalysis, Colaris, Melaris test 제품들이 있다. 최근에는 microarray 방법 또한 멘델리안 질환, 또는 서서히 진행되는 질환의 copy number variation을 detection 하는 방법으로 널리 이용되고 있다. NGS 플랫폼의 경우, 원하는 유전자, 또는 일부 영역만을 타겟하여 그 부분만 시퀀싱하는 targeted resequencing 방법이 선호되는 추세이다. Targeted resequencing은 타겟 영역만 캡처하고, 증폭 후 시퀀싱 하는 방법인데, 적은 영역만을 해독하므로 시퀀싱 비용이 적게 들고, 데이터 용량 또한 적어 분석이 용이하기 때문에 bench top sequencer를 이용할 수 있어 비용 대비 효과가 큰 것으로 알려져 있다. 향후 Sanger sequencing이나 microarray보다 진단에 많이 이용될 것으로 기대된다. | |
| − | + | | |
| − | + | | |
| − | | + | |
| − | + | '''임상에 NGS를 이용할 경우 장점'''은 3가지로 나눌 수 있다. | |
| − | | + | |
| − | + | 첫번째는 '''민감도(sensitivity)'''이다. NGS를 이용할 경우, 타겟 영역의 모든 nucleotide 정보를 알 수 있다는 장점이 있다. 이 때, 해독배수를 높일수록 희귀 변이를 찾아낼 수 있는 확률이 더욱 커지게 된다. Choi 등은 바터 증후군(Barter syndrome) 환자의 샘플을 99X로 whole exome sequencing을 진행한 결과, SNP genotyping으로는 찾아낼 수 없었던 SLC26A3 유전자의 rare missense variant를 발견하였다고 보고하였다. | |
| − | | + | |
| − | + | 두번째 장점으로는 '''유전자의 발현 등 환경에 따른 변화 정도를 알 수 있다'''는 것이다. Probe를제작한 후 진행되는 Microarray 방법과는 달리, NGS를 이용하면 예상하지 못한 염색체 이상 등을 발견할 수 있다는 장점이 있다. NGS로 알아낸 변이 정보를 Sanger sequencing을 이용하여 간단히 검증하여 정확도를 높일 수 있다. 특히, 암조직의 경우, gene fusion이 일어났는지 여부 까지도 알 수 있다는 장점이 있다. | |
| − | | + | |
| − | + | 세번째 장점으로는 '''샘플 필요량이 적다'''는 것이다. Sanger sequencing 방법의 경우 최소 1ug 이상의 DNA가 필요하며, cytogenetic assay의 경우도 최소 250ng의 DNA가 필요하다. 그러나 Ion Torrent PGM의 경우 50ng 이하의 DNA로도 시퀀싱이 가능하며, 재연성 또한 높기 때문에 진단에 활용할 경우 장점이 될 수 있다. | |
| − | + | | |
| − | | + | |
| − | + | 그러나'''NGS를 임상에 활용하기 위해서는 해결해야 할 문제'''가 남아있다. | |
| − | | + | |
| − | + | 첫째로 '''데이터 분석 문제'''이다. 수십, 수백 Gb나 되는 데이터를 정확히 빠른 시간 내에 분석해 내기 위해서는 여러 분석 도구들을 이용할 줄 알아야 한다. 여러 연구실에서 open source tool들을 이용하여 분석하고 있지만, 진단에 이용하기 위해서는 보다 체계화 되고 검증된 도구가 필요하다. | |
| − | | + | |
| − | + | 두번째는 '''컴퓨팅 파워'''이다. NGS 데이터를 다루기 위해서는 최소한 8 quad core, 32Gb RAM, 10 terabytes의 disk space가 필요하며, 숙련된 IT 전문가와 bioinformatics 스탭이 필요하다. 이러한 환경을 갖추기 위해서는 수천 달러를 투자해야 하기 때문에, NGS의 임상 적용을 위해 환경이 잘 갖추어진 기관과 협력하는 편이 더 나을 수 있다. | |
| − | | + | |
| − | + | 세번째 문제점은 '''법적, 윤리적 이슈'''이다. 개인의 유전정보를 공개해도 되는지 여부에 대해 여러 이슈들이 존재해 왔으며, 아직도 해결되지 못한 과제이다. 개인유전정보가 공개될 경우, 보험 가입이 거부당하거나, 법적 분쟁, 또는 고용의 불평등이 존재할 수 있기 때문이다. 그러나 이러한 문제점들은 게놈 전문가와 임상의들과의 협력을 통하여 해결될 수 있다. 가장 이상적인 것은 개인유전정보 서비스를 받은 고객에게서 변이가 발견될 경우, 게놈 분석 기관에서 의사에게 통보 후, 의사가 환자 개인에게 상담하는 것이다. | |
| − | | + | |
| − | + | NGS의 임상 적용의 장단점, 문제점을 알아보았다. 현재의 NGS 기술로 임상 적용이 가능하게 되었으며, 더 정확한 진단과 치료를 하기 위해 게놈 연구자와 임상의, 판매 기업들의 협조가 매우 필요한 시점이다. | |
| − | + | | |
| − | + | | |
| − | + | '''참고문헌''' | |
| − | http://www.ncbi.nlm.nih.gov/pubmed/22375550 | + | Next-generation sequencing: ready for theclinics? |
| + | |||
| + | [http://www.ncbi.nlm.nih.gov/pubmed/22375550 http://www.ncbi.nlm.nih.gov/pubmed/22375550] | ||
Latest revision as of 08:32, 12 October 2022
NGS 기술의 현재: NGS 기술의 임상 적용 Yes or No?
인간게놈프로젝트(human genome project, HGP)의 완료로 30억 쌍의 인간 DNA 염기가 해독됨에 따라, 인간 게놈으로부터 얻은 정보를 진단과 치료에 활용하기 위한 노력이 계속되고 있다. 드물긴 하지만, 현재도 유전 질환의 진단에 활용하고, 유전형에 맞추어 치료방법을 선택하는 몇몇 사례가 보고되었다. 이는 post-HGP와 대용량 데이터 처리기술의 발달이 이뤄 낸 성과이다.
차세대 게놈 분석기가 최초로 도입된 2005년 이래, 끊임없는 발전을 거듭하여 2012년 현재는 데이터 생산량이 확연히 늘고, 에러율이 줄어들었을 뿐 아니라, 다양한 사이즈의 라이브러리 제작이 가능하게 되었다. 결과, reference 서열이 없이도 분석 가능한 de novo sequencing, 코딩 영역만 해독하는 exome sequencing, 유전자의 발현 변화를 볼 수 있는 transcriptome profiling, 질병의 진행여부 등 환경 변화에 따른 메틸화 정도를 알 수 있는 methylation profiling, 토양이나 물 등의 환경 시료를 이용하는 metagenome 연구까지도 NGS를 이용하여 가능하게 되었다. 게다가, 연구를 넘어서 진단에까지 이용되고 있는데, 특히, NGS를 이용한 암진단 사례가 급속히 늘고 있다.
Welch 등은 전장유전체 분석을 이용하여 oncogene의 fusion이 일어났음을 알아내어 stem cell plantation 치료 대신 화학요법 치료로 방향을 전환할 수 있었음을 보고하였다.
또한 전장유전체 분석을 이용하여 환자의 정상 피부와 골수(leukemia)를 이용하여 초기 유방암과 자궁암의 진단에 활용한 예가 있다. 이 환자의 경우, 피부 세포에 TP53 유전자의 exon 7-9의 novel한 3Kb heterozygous deletion이 있으며, leukemia 세포에 같은 영역의 homozygous deletion이 있음을 시퀀싱을 통해 발견하였고, 이 변이에 의해 암에 걸렸을 확률이 높다는 것을 발견하였다.
Mellman 등은 Ion Torrent PGM machine을 이용하여 전염성이 강한 Shiga toxin을 생산하는 E.coli O104:H4를 10일 만에 동정하여 발표함으로써 독일에서 발생한 식중독 원인균을 찾아내는 데 크게 공헌하여 화제가 된 바 있다.
위와 같이 진단 등에 NGS를 활용할 수 있게 된 이유는 NGS 기술이 끊임없이 향상되고, 시퀀싱 단가가 줄어들었기 때문이다. 특히 MiSeq, 454 GS Junior, Ion Torrent PGM과 같은 소형 해독기(bench top sequencer)의 출현으로 인해 가능해 진 것이다.
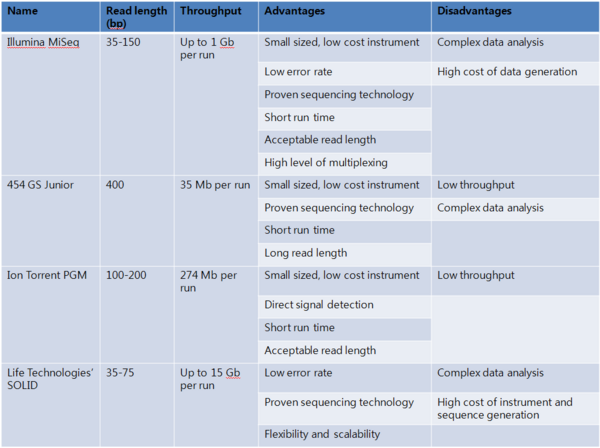
다음 표는 bench top sequencer들의 장점과 단점을 요약한 것이다.
고전적으로 Sanger sequencing과 PCR 방법은 암 및 감염성 질환의 유전 변이를 진단하는 도구로 이용되어 왔으며, 그 예로 Myraid Genetic Laboratory사의 BRACAnalysis, Colaris, Melaris test 제품들이 있다. 최근에는 microarray 방법 또한 멘델리안 질환, 또는 서서히 진행되는 질환의 copy number variation을 detection 하는 방법으로 널리 이용되고 있다. NGS 플랫폼의 경우, 원하는 유전자, 또는 일부 영역만을 타겟하여 그 부분만 시퀀싱하는 targeted resequencing 방법이 선호되는 추세이다. Targeted resequencing은 타겟 영역만 캡처하고, 증폭 후 시퀀싱 하는 방법인데, 적은 영역만을 해독하므로 시퀀싱 비용이 적게 들고, 데이터 용량 또한 적어 분석이 용이하기 때문에 bench top sequencer를 이용할 수 있어 비용 대비 효과가 큰 것으로 알려져 있다. 향후 Sanger sequencing이나 microarray보다 진단에 많이 이용될 것으로 기대된다.
임상에 NGS를 이용할 경우 장점은 3가지로 나눌 수 있다.
첫번째는 민감도(sensitivity)이다. NGS를 이용할 경우, 타겟 영역의 모든 nucleotide 정보를 알 수 있다는 장점이 있다. 이 때, 해독배수를 높일수록 희귀 변이를 찾아낼 수 있는 확률이 더욱 커지게 된다. Choi 등은 바터 증후군(Barter syndrome) 환자의 샘플을 99X로 whole exome sequencing을 진행한 결과, SNP genotyping으로는 찾아낼 수 없었던 SLC26A3 유전자의 rare missense variant를 발견하였다고 보고하였다.
두번째 장점으로는 유전자의 발현 등 환경에 따른 변화 정도를 알 수 있다는 것이다. Probe를제작한 후 진행되는 Microarray 방법과는 달리, NGS를 이용하면 예상하지 못한 염색체 이상 등을 발견할 수 있다는 장점이 있다. NGS로 알아낸 변이 정보를 Sanger sequencing을 이용하여 간단히 검증하여 정확도를 높일 수 있다. 특히, 암조직의 경우, gene fusion이 일어났는지 여부 까지도 알 수 있다는 장점이 있다.
세번째 장점으로는 샘플 필요량이 적다는 것이다. Sanger sequencing 방법의 경우 최소 1ug 이상의 DNA가 필요하며, cytogenetic assay의 경우도 최소 250ng의 DNA가 필요하다. 그러나 Ion Torrent PGM의 경우 50ng 이하의 DNA로도 시퀀싱이 가능하며, 재연성 또한 높기 때문에 진단에 활용할 경우 장점이 될 수 있다.
그러나NGS를 임상에 활용하기 위해서는 해결해야 할 문제가 남아있다.
첫째로 데이터 분석 문제이다. 수십, 수백 Gb나 되는 데이터를 정확히 빠른 시간 내에 분석해 내기 위해서는 여러 분석 도구들을 이용할 줄 알아야 한다. 여러 연구실에서 open source tool들을 이용하여 분석하고 있지만, 진단에 이용하기 위해서는 보다 체계화 되고 검증된 도구가 필요하다.
두번째는 컴퓨팅 파워이다. NGS 데이터를 다루기 위해서는 최소한 8 quad core, 32Gb RAM, 10 terabytes의 disk space가 필요하며, 숙련된 IT 전문가와 bioinformatics 스탭이 필요하다. 이러한 환경을 갖추기 위해서는 수천 달러를 투자해야 하기 때문에, NGS의 임상 적용을 위해 환경이 잘 갖추어진 기관과 협력하는 편이 더 나을 수 있다.
세번째 문제점은 법적, 윤리적 이슈이다. 개인의 유전정보를 공개해도 되는지 여부에 대해 여러 이슈들이 존재해 왔으며, 아직도 해결되지 못한 과제이다. 개인유전정보가 공개될 경우, 보험 가입이 거부당하거나, 법적 분쟁, 또는 고용의 불평등이 존재할 수 있기 때문이다. 그러나 이러한 문제점들은 게놈 전문가와 임상의들과의 협력을 통하여 해결될 수 있다. 가장 이상적인 것은 개인유전정보 서비스를 받은 고객에게서 변이가 발견될 경우, 게놈 분석 기관에서 의사에게 통보 후, 의사가 환자 개인에게 상담하는 것이다.
NGS의 임상 적용의 장단점, 문제점을 알아보았다. 현재의 NGS 기술로 임상 적용이 가능하게 되었으며, 더 정확한 진단과 치료를 하기 위해 게놈 연구자와 임상의, 판매 기업들의 협조가 매우 필요한 시점이다.
참고문헌
Next-generation sequencing: ready for theclinics?