From PGI
(Created page with "= '''생명정보 데이터 분석 방법의 현재''' = NGS의 활용분야는 점차 다양해져, DNA sequencing 분석으로 SNPs(Single Nucleotide Polymorp...") |
|||
| Line 1: | Line 1: | ||
| − | |||
| − | + | = '''생명정보 데이터 분석 방법의 현재''' = | |
| − | |||
| − | |||
| − | DNA sequencing | + | NGS의 활용분야는 점차 다양해져, DNA sequencing 분석으로 SNPs(Single Nucleotide Polymorphism) 및 CNVs(Copy Number Variations) detection, RNA sequencing 분석을 통한 transctiptomes 또는 정량분석, 후성유전체 분석 등의 방법이 있다. |
| − | + | | |
| − | | + | DNA sequencing raw read의 분석 과정은 크게 두 가지 방법으로 나눌 수 있다. 첫 번째는 raw read의 mapping과 assembling, quality control, quality score re-calibration, genome의 '''“difficult” region의 realignment'''이다. 두 번째는'' '''''variant calling(SNP, InDels, CNVs) 및 annotation'''과정이다. |
| − | + | | |
| − | + | 본 리뷰에서는 이 두 과정에 주로 사용되고 있는 software들에 대해 알아본다. | |
| − | + | | |
| − | + | | |
| − | + | === 1. Alignment === | |
| − | |||
| − | |||
| − | + | DNA sequencing 데이터 분석의 첫 단계는 '''alignment와 assembly'''이다. Alignment 단계는 DNA-Seq reads를 reference genome에 mapping하는 단계이다. | |
| − | + | | |
| − | + | '''Alignment'''에 주로 이용되는 알고리즘은 크게 <u>hash-based 방법과 Burrow-Wheeler Transform 방법</u>의 두 종류이다. Hash-based 알고리즘 중 input reads의 set에 hash table을 구축하는 방법을 이용하는 software인 MAQ, ELAND(Illumina의 알고리즘), SHRiMP, ZOOM이 있으며, reference genome의 set에 hash table을 구축하는 방법을 이용하는 software로 SOAPv2, BFAST, MOSAIK, Novoalign, PERM이 있다. 최근에 많이 쓰이는 알고리즘은 Burrow-Wheeler Transform(BWT)을 사용하여 string matching을 하는 방법으로, 대표적인 software로는 BOWTIE, BWA, SOAPv2가 있다. | |
| − | |||
| − | |||
| − | + | === 2. Assembly === | |
| − | |||
| − | |||
| − | + | <u>Reference genome이 존재하지 않는 생물 종을 분석할 경우</u>는 '''de novo assembly'''를 해야만 한다. De novo assembly 방법은 <u>overlap graph 알고리즘과 de Bruijn graph 알고리즘을 이용하는 방법</u>의 두 가지로 나뉜다. Overlap graph를 이용하는 software로는 Celera Assembler 또는 Arachne가 있다. | |
| − | | + | |
| − | + | NGS data assembly에 사용되는 tool들은 대부분 De Bruijn geaph 방식을 사용한다. De Bruijn graph 방식을 이용하는 software는 Velvet, SOAPdeNOVO, ABySS가 있다. De Bruijn graph 방법의 선택 옵션 중 하나인 k-mer size는 매우 중요한 옵션으로, k-mer size의 조건에 따라 assembly sequencing error가 달라질 수 있다. | |
| − | | + | |
| − | + | === 3. 리드 품질관리 === | |
| − | |||
| − | |||
| − | | + | NGS 데이터는 short reads를 alignment해서 사용하는 것이기 때문에, 리드 품질관리의 이슈는 초창기부터 계속되어 왔다. Reads의 alignment와 assembly를 한 후, 다음 단계가 quality control(QC) 단계이다. |
| − | + | | |
| − | < | + | '''Basic QC'''의 첫 번째 단계는 <u>aligned reads를 SAM 또는 BAM 포맷의 파일로 전환</u>하는 것이다. 이 단계의 output은 깨끗한 리드들이 sorting 되고, indexing 된 BAM 형태이며, 다음 단계인 '''Advanced QC''' 단계로 진행된다. Misaligned reads와 퀄리티가 낮은 리드들은 SNP 발굴과 genotyping 단계에 많은 영향을 미치기 때문에, NGS 데이터의 신뢰도 측면에서 advanced QC 단계는 매우 중요하다. Advanced QC 단계에 주로 사용되는 software로는 SAMTOOLS, PICARD, GATK가 있다. |
| − | + | | |
| − | + | === 4. Variant calling and Annotation === | |
| − | |||
| − | |||
| − | ''' | + | Variant calling에 주로 사용되는 방법은 3가지로 나눌 수 있다. 첫 번째 방법은 '''Sequence Variant Analyzer(SVA)'''를 이용하는 방법인데, SVA 버전은 ENSEMBL hg18 annotations에 링크가 되어 있다. 두 번째 방법은'''SAMTOOLS'''로 variant detection을 한 후, VCF 파일로 export 한다. 그 다음, ANNOVAR를 사용하여 annotation하는 것이다. 최근 버전의 ANNOVAR는 1000 Genomes Project의 정보와 Complete Genomics에서 얻은 60 genomes 정보를 annotation 할 수 있다. 세 번째 방법은 '''GATK suite의 UnifiedGenotyper(V2)'''를 이용하여 SNP 및 Indel을 감지한 후, VCF로 export 한다. 그 다음, ANNOVAR로 annotation 하는 방법이다. |
| − | + | | |
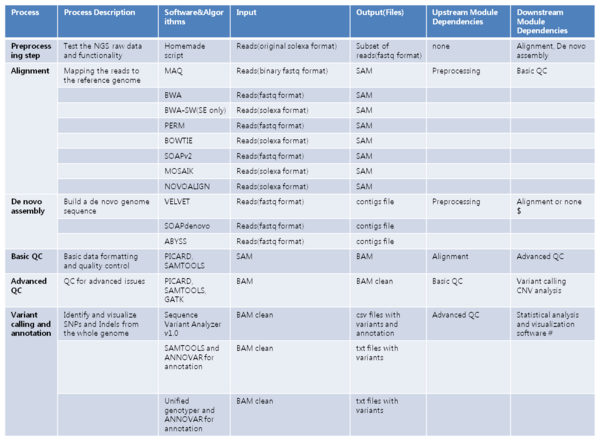
| − | 편집 : Park.HJ | + | <span style="line-height: 1.5em;">[[File:NGS분석5.png|center|600px|NGS분석5.png]] Table 1. NGS pipeline 리뷰</span> |
| + | |||
| + | | ||
| + | |||
| + | | ||
| + | |||
| + | '''참고자료''' | ||
| + | |||
| + | :Next generation sequence analysis and computational genomics using graphical pipeline workflows. Genes (Basel). 2012. | ||
| + | |||
| + | '''저자''' | ||
| + | |||
| + | 글 : Lee.SY | ||
| + | |||
| + | 편집 : Park.HJ | ||
키워드 : NGS, annotation, variant calling, assembly, SAMTOOLS, Bruijn graph, SOAPdeNOVO, ABySS, ANNOVAR, alignment 등 | 키워드 : NGS, annotation, variant calling, assembly, SAMTOOLS, Bruijn graph, SOAPdeNOVO, ABySS, ANNOVAR, alignment 등 | ||
Latest revision as of 08:31, 12 October 2022
Contents
생명정보 데이터 분석 방법의 현재
NGS의 활용분야는 점차 다양해져, DNA sequencing 분석으로 SNPs(Single Nucleotide Polymorphism) 및 CNVs(Copy Number Variations) detection, RNA sequencing 분석을 통한 transctiptomes 또는 정량분석, 후성유전체 분석 등의 방법이 있다.
DNA sequencing raw read의 분석 과정은 크게 두 가지 방법으로 나눌 수 있다. 첫 번째는 raw read의 mapping과 assembling, quality control, quality score re-calibration, genome의 “difficult” region의 realignment이다. 두 번째는 variant calling(SNP, InDels, CNVs) 및 annotation과정이다.
본 리뷰에서는 이 두 과정에 주로 사용되고 있는 software들에 대해 알아본다.
1. Alignment
DNA sequencing 데이터 분석의 첫 단계는 alignment와 assembly이다. Alignment 단계는 DNA-Seq reads를 reference genome에 mapping하는 단계이다.
Alignment에 주로 이용되는 알고리즘은 크게 hash-based 방법과 Burrow-Wheeler Transform 방법의 두 종류이다. Hash-based 알고리즘 중 input reads의 set에 hash table을 구축하는 방법을 이용하는 software인 MAQ, ELAND(Illumina의 알고리즘), SHRiMP, ZOOM이 있으며, reference genome의 set에 hash table을 구축하는 방법을 이용하는 software로 SOAPv2, BFAST, MOSAIK, Novoalign, PERM이 있다. 최근에 많이 쓰이는 알고리즘은 Burrow-Wheeler Transform(BWT)을 사용하여 string matching을 하는 방법으로, 대표적인 software로는 BOWTIE, BWA, SOAPv2가 있다.
2. Assembly
Reference genome이 존재하지 않는 생물 종을 분석할 경우는 de novo assembly를 해야만 한다. De novo assembly 방법은 overlap graph 알고리즘과 de Bruijn graph 알고리즘을 이용하는 방법의 두 가지로 나뉜다. Overlap graph를 이용하는 software로는 Celera Assembler 또는 Arachne가 있다.
NGS data assembly에 사용되는 tool들은 대부분 De Bruijn geaph 방식을 사용한다. De Bruijn graph 방식을 이용하는 software는 Velvet, SOAPdeNOVO, ABySS가 있다. De Bruijn graph 방법의 선택 옵션 중 하나인 k-mer size는 매우 중요한 옵션으로, k-mer size의 조건에 따라 assembly sequencing error가 달라질 수 있다.
3. 리드 품질관리
NGS 데이터는 short reads를 alignment해서 사용하는 것이기 때문에, 리드 품질관리의 이슈는 초창기부터 계속되어 왔다. Reads의 alignment와 assembly를 한 후, 다음 단계가 quality control(QC) 단계이다.
Basic QC의 첫 번째 단계는 aligned reads를 SAM 또는 BAM 포맷의 파일로 전환하는 것이다. 이 단계의 output은 깨끗한 리드들이 sorting 되고, indexing 된 BAM 형태이며, 다음 단계인 Advanced QC 단계로 진행된다. Misaligned reads와 퀄리티가 낮은 리드들은 SNP 발굴과 genotyping 단계에 많은 영향을 미치기 때문에, NGS 데이터의 신뢰도 측면에서 advanced QC 단계는 매우 중요하다. Advanced QC 단계에 주로 사용되는 software로는 SAMTOOLS, PICARD, GATK가 있다.
4. Variant calling and Annotation
Variant calling에 주로 사용되는 방법은 3가지로 나눌 수 있다. 첫 번째 방법은 Sequence Variant Analyzer(SVA)를 이용하는 방법인데, SVA 버전은 ENSEMBL hg18 annotations에 링크가 되어 있다. 두 번째 방법은SAMTOOLS로 variant detection을 한 후, VCF 파일로 export 한다. 그 다음, ANNOVAR를 사용하여 annotation하는 것이다. 최근 버전의 ANNOVAR는 1000 Genomes Project의 정보와 Complete Genomics에서 얻은 60 genomes 정보를 annotation 할 수 있다. 세 번째 방법은 GATK suite의 UnifiedGenotyper(V2)를 이용하여 SNP 및 Indel을 감지한 후, VCF로 export 한다. 그 다음, ANNOVAR로 annotation 하는 방법이다.
Table 1. NGS pipeline 리뷰
참고자료
- Next generation sequence analysis and computational genomics using graphical pipeline workflows. Genes (Basel). 2012.
저자
글 : Lee.SY
편집 : Park.HJ
키워드 : NGS, annotation, variant calling, assembly, SAMTOOLS, Bruijn graph, SOAPdeNOVO, ABySS, ANNOVAR, alignment 등