From PGI
(Created page with "= '''유전적 심장질환을 임상적 분자진단에 이용하는 Bench-Top 게놈 해독기 비교''' = 분자진단은 유전 질환의 관리에서 진...") |
|||
| Line 1: | Line 1: | ||
| − | |||
| − | + | = '''유전적 심장질환을 임상적 분자진단에 이용하는 Bench-Top 게놈 해독기 비교''' = | |
| − | |||
| − | |||
| − | + | 분자진단은 유전 질환의 관리에서 진단과 효과적인 치료를 추천하지만, 비용과 시간, 서비스의 유효성의 문제들로 인해 많은 시도가 필요하다. 차세대 해독기술을 이용한 DNA 분석은 이러한 문제들을 잠재적으로 극복할 수 있다. 부정맥 증후군이나 심근증과 같은 유전적 심장질환은 임상적으로 이용하기 위해서는 NGS 분석의 진화에 적용될 수 있다. | |
| − | |||
| − | |||
| − | + | [[File:Bench-Top 01.png|center|500.px]] | |
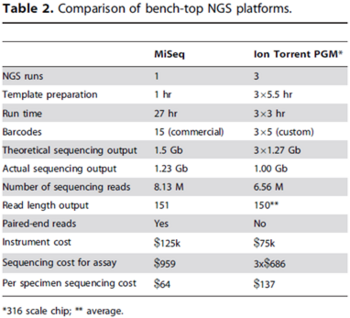
| − | + | 대다수 bench-top NGS 플랫폼들은 MiSeq과 Ion Torrent Personal Genome Machine을 포함하여 Gb 규모의 DNA 해독에 27시간 이하의 짧은 시간이 소요된다. 초기의 연구는 임상적 유효한 유전적 타겟의 특징을 이용하였다. 예를 들어 박테리아 게놈이나 인간의 유방암 BRCA 유전자, 세포섬유종 CFTR 유전자, 암의 체세포 변이 HLA 유형 등이 있다. 임상 진단에서 높은 분석 생산량과 빠른 속도는 매력적이다. 따라서 본 논문에서는 MiSeq과 Ion Torrent PGM DNA 해독 플랫폼을 비교하여 임상적 진단에 이용하는 정교한 파이프라인의 진화에 대한 정보를 제공하였다. | |
| − | |||
| − | |||
| − | + | 익명의 영국인 15명 DNA 샘플을 추출하였고, 그 중 11명에서는 생어시퀀싱으로 Long QT syndrome 관련 유전자 변이 5개가 공통적으로 확인되었고, 나머지 4명은 확인된 5개 유전변이를 엑솜시퀀싱으로 확인하였다. 부정맥에 관련이 있는 KCNQ1, KCNH2, SCN5A, KCNE1, KCNE2, RYR2 총 6개 유전자를 타겟하여 PCR 증폭하였다. 그 다음 플랫폼에 맞는 키트를 사용하여 MiSeq과 Ion Torrent PGM 시퀀싱을 수행하였다. 해독된 데이터는 FastQC version 0.9.5를 이용하여 각 샘플의 퀄리티를 측정하였고, BWA를 이용하여 alignment하였으며, GATK, Picard를 이용하여 변이를 감지하였다. | |
| − | |||
| − | |||
| − | + | [[File:Bench-Top 02.png|center|350px|Bench-Top 02.png]] | |
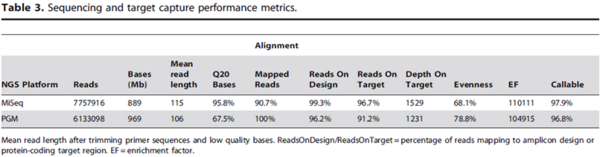
| − | [[ | + | [[File:Bench-Top 03.png|center|600px|Bench-Top 03.png]] <br/> MiSeq은 총 1,230Mb를 생산하였으며, Ion Torrent PGM(316chip)은 총 1,001Mb를 생산하였다. 또 높은 퀄리티의 데이터(Q20)를 MiSeq은 95.8%, PGM은 67.5% 생산하였다. 6가지 유전자 전반적으로 98.8%가 매핑되었으나, 그 중 SCN5A1, KCNE1, RYR2는 100% 매핑되었고, KCNQ1과 KCNH2는 MiSeq에서 96.2%와 94.1%, PGM에서는 93.1%와 88.9% 매핑되었다. |
| − | |||
| − | |||
| − | + | [[File:Bench-Top 04.png|center|600px|Bench-Top 04.png]] | |
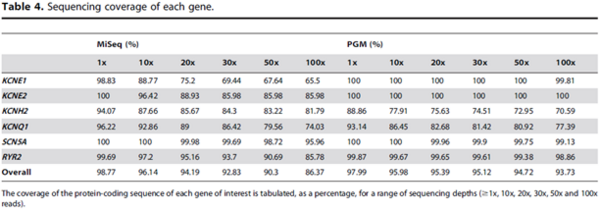
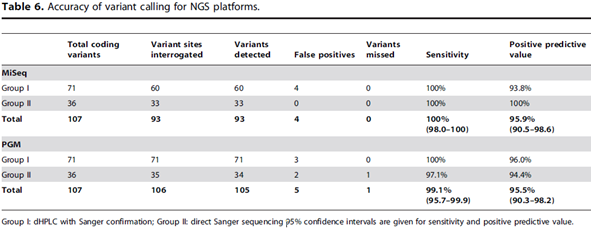
| − | + | dHPLC 변이 검사에 의해 식별된 그룹과 생어시퀀싱으로 식별된 그룹으로 샘플을 나누어 진행하였으며, NGS는 생어보다 초기 단계에서 변이를 감지하고 이전에 감지되지 않았던 변이를 감지할 수 있었다. 정확도도 MiSeq은 95.9%, PGM은 95.4%로 높게 나타났으며, 위 표는 각 유전자별 해독 배수에 따른 정확도를 나타내었다. 특히 KCNE1은 12개 샘플에서 확인되었는데, MiSeq에서는 chr21 35821821과 chr21 35821795로 단일 변이들이 발견되었으나, PGM에서는 발견되지 않았다. 또한 PGM은 KCNH2 유전자의 loci 2곳(chr7 150645534와 chr11 2594088)을 감지하지 못했다. | |
| − | |||
| − | |||
| − | + | [[File:Bench-Top 05.png|center|600px|Bench-Top 05.png]] | |
| − | + | 비용과 시간적인 측면에서 MiSeq은 959달러의 비용으로 한 번 실험을 수행하였고, PGM은 686달러의 비용으로 여러 번 실험된 것이다. 그러나 MiSeq은 28시간 동안 pooling과 sequencing이 이루어졌고, PGM은 시간이 단축되었으나 DNA 농축 등의 시간이 추가로 요구되었다. 요약하면 MiSeq은 100%의 민감도와 95.9%의 정확도이며, PGM은 99.1%의 민감도와 95.5%의 정확도를 보인다. | |
| − | |||
| − | |||
| − | === | + | === 맺음말 === |
| − | |||
| − | |||
| − | + | 분자 유전학적 검사는 유전적 심장질환의 진단을 위해 예후와 치료를 안내할 수 있지만, 비용이 높아 현실적인 문제가 있었다. 분자진단을 위해 NGS 플랫폼을 이용하여 유전적 부정맥 증후군과 관련된 6개 유전자영역을 MiSeq과 PGM 그리고 PCR을 이용하여 15명의 환자의 게놈을 해독하였다. MiSeq에서 97.9%, PGM에서 96.8%의 정확성을 나타내었으나, GC가 누락되는 등 에러율 문제가 남아 있었다. 총 107개 변이를 생어시퀀싱하여 두 플랫폼을 평가하였으며, 민감도는 MiSeq 100%, PGM 99.1%로 MiSeq이 높았으나, PGM이 저렴하고 빠르다는 장점이 있었다. 반면 MiSeq은 생산량이 많고, 실험 방법이 간단하다는 장점이 있었다. 생어시퀀싱에 비해 두 플랫폼은 상당한 비용과 시간이 절감되는 효과를 기대해 볼 수 있다. MiSeq과 PGM 모두 PCR 기반의 분자진단 플랫폼으로 정확한 변이를 감지할 수 있고, 유전적 심장질환의 분자진단을 위한 플랫폼으로서의 가능성이 보이는 결과를 도출했다. 성능의 차이가 있었으나, 비용이나 프로토콜의 안정성과 사용의 용이성 측면에서 기존의 방식보다 빠르고 저렴하면서도 포괄적인 결과를 보여주었다. | |
| − | |||
| − | + | ||
| + | === 참고문헌 === | ||
| + | |||
| + | |||
| + | |||
| + | Towards Clinical Molecular Diagnosis of Inherited Cardiac Conditions: A Comparison of Bench-Top Genome DNA Sequencers | ||
| + | |||
| + | |||
| + | |||
| + | === 역저자 === | ||
| + | |||
| + | |||
| + | |||
| + | 글 : Park.HyeonJi | ||
| + | |||
| + | 편집 : Jeon.EunSook, Ahn.Kung | ||
Latest revision as of 11:16, 11 October 2022
유전적 심장질환을 임상적 분자진단에 이용하는 Bench-Top 게놈 해독기 비교
분자진단은 유전 질환의 관리에서 진단과 효과적인 치료를 추천하지만, 비용과 시간, 서비스의 유효성의 문제들로 인해 많은 시도가 필요하다. 차세대 해독기술을 이용한 DNA 분석은 이러한 문제들을 잠재적으로 극복할 수 있다. 부정맥 증후군이나 심근증과 같은 유전적 심장질환은 임상적으로 이용하기 위해서는 NGS 분석의 진화에 적용될 수 있다.
대다수 bench-top NGS 플랫폼들은 MiSeq과 Ion Torrent Personal Genome Machine을 포함하여 Gb 규모의 DNA 해독에 27시간 이하의 짧은 시간이 소요된다. 초기의 연구는 임상적 유효한 유전적 타겟의 특징을 이용하였다. 예를 들어 박테리아 게놈이나 인간의 유방암 BRCA 유전자, 세포섬유종 CFTR 유전자, 암의 체세포 변이 HLA 유형 등이 있다. 임상 진단에서 높은 분석 생산량과 빠른 속도는 매력적이다. 따라서 본 논문에서는 MiSeq과 Ion Torrent PGM DNA 해독 플랫폼을 비교하여 임상적 진단에 이용하는 정교한 파이프라인의 진화에 대한 정보를 제공하였다.
익명의 영국인 15명 DNA 샘플을 추출하였고, 그 중 11명에서는 생어시퀀싱으로 Long QT syndrome 관련 유전자 변이 5개가 공통적으로 확인되었고, 나머지 4명은 확인된 5개 유전변이를 엑솜시퀀싱으로 확인하였다. 부정맥에 관련이 있는 KCNQ1, KCNH2, SCN5A, KCNE1, KCNE2, RYR2 총 6개 유전자를 타겟하여 PCR 증폭하였다. 그 다음 플랫폼에 맞는 키트를 사용하여 MiSeq과 Ion Torrent PGM 시퀀싱을 수행하였다. 해독된 데이터는 FastQC version 0.9.5를 이용하여 각 샘플의 퀄리티를 측정하였고, BWA를 이용하여 alignment하였으며, GATK, Picard를 이용하여 변이를 감지하였다.
MiSeq은 총 1,230Mb를 생산하였으며, Ion Torrent PGM(316chip)은 총 1,001Mb를 생산하였다. 또 높은 퀄리티의 데이터(Q20)를 MiSeq은 95.8%, PGM은 67.5% 생산하였다. 6가지 유전자 전반적으로 98.8%가 매핑되었으나, 그 중 SCN5A1, KCNE1, RYR2는 100% 매핑되었고, KCNQ1과 KCNH2는 MiSeq에서 96.2%와 94.1%, PGM에서는 93.1%와 88.9% 매핑되었다.
dHPLC 변이 검사에 의해 식별된 그룹과 생어시퀀싱으로 식별된 그룹으로 샘플을 나누어 진행하였으며, NGS는 생어보다 초기 단계에서 변이를 감지하고 이전에 감지되지 않았던 변이를 감지할 수 있었다. 정확도도 MiSeq은 95.9%, PGM은 95.4%로 높게 나타났으며, 위 표는 각 유전자별 해독 배수에 따른 정확도를 나타내었다. 특히 KCNE1은 12개 샘플에서 확인되었는데, MiSeq에서는 chr21 35821821과 chr21 35821795로 단일 변이들이 발견되었으나, PGM에서는 발견되지 않았다. 또한 PGM은 KCNH2 유전자의 loci 2곳(chr7 150645534와 chr11 2594088)을 감지하지 못했다.
비용과 시간적인 측면에서 MiSeq은 959달러의 비용으로 한 번 실험을 수행하였고, PGM은 686달러의 비용으로 여러 번 실험된 것이다. 그러나 MiSeq은 28시간 동안 pooling과 sequencing이 이루어졌고, PGM은 시간이 단축되었으나 DNA 농축 등의 시간이 추가로 요구되었다. 요약하면 MiSeq은 100%의 민감도와 95.9%의 정확도이며, PGM은 99.1%의 민감도와 95.5%의 정확도를 보인다.
맺음말
분자 유전학적 검사는 유전적 심장질환의 진단을 위해 예후와 치료를 안내할 수 있지만, 비용이 높아 현실적인 문제가 있었다. 분자진단을 위해 NGS 플랫폼을 이용하여 유전적 부정맥 증후군과 관련된 6개 유전자영역을 MiSeq과 PGM 그리고 PCR을 이용하여 15명의 환자의 게놈을 해독하였다. MiSeq에서 97.9%, PGM에서 96.8%의 정확성을 나타내었으나, GC가 누락되는 등 에러율 문제가 남아 있었다. 총 107개 변이를 생어시퀀싱하여 두 플랫폼을 평가하였으며, 민감도는 MiSeq 100%, PGM 99.1%로 MiSeq이 높았으나, PGM이 저렴하고 빠르다는 장점이 있었다. 반면 MiSeq은 생산량이 많고, 실험 방법이 간단하다는 장점이 있었다. 생어시퀀싱에 비해 두 플랫폼은 상당한 비용과 시간이 절감되는 효과를 기대해 볼 수 있다. MiSeq과 PGM 모두 PCR 기반의 분자진단 플랫폼으로 정확한 변이를 감지할 수 있고, 유전적 심장질환의 분자진단을 위한 플랫폼으로서의 가능성이 보이는 결과를 도출했다. 성능의 차이가 있었으나, 비용이나 프로토콜의 안정성과 사용의 용이성 측면에서 기존의 방식보다 빠르고 저렴하면서도 포괄적인 결과를 보여주었다.
참고문헌
Towards Clinical Molecular Diagnosis of Inherited Cardiac Conditions: A Comparison of Bench-Top Genome DNA Sequencers
역저자
글 : Park.HyeonJi
편집 : Jeon.EunSook, Ahn.Kung